Investigadores de la Universidad de Oregon, Estados Unidos, desarrollaron un método para predecir la fenología de la vid, es decir, los momentos clave del desarrollo de la uva, tales como el brote, la floración y el envero. El trabajo que a continuación publicamos propone una solución que combina modelos biofísicos tradicionales con técnicas avanzadas de aprendizaje automático. El objetivo es mejorar la precisión de las predicciones y facilitar la toma de decisiones en viñedos, como la programación del riego o la fertilización.

Un grupo de investigadores ha presentado un nuevo método para predecir la fenología de la vid, es decir, los momentos clave del desarrollo de la uva como el brote, la floración y el envero. El trabajo, realizado por William Solow y Sandhya Saisubramanian, propone una solución que combina modelos biofísicos tradicionales con técnicas avanzadas de aprendizaje automático.
La predicción precisa de la fenología es fundamental para los viticultores. Un error en el cálculo de estos momentos puede afectar tanto al rendimiento como a la calidad de la cosecha. Hasta ahora, los modelos más utilizados se basan en datos históricos y en variables como la temperatura acumulada (modelo GDD), pero suelen ser poco flexibles y no consideran otros factores climáticos relevantes. Por otro lado, los métodos basados en inteligencia artificial requieren grandes cantidades de datos, algo que no siempre está disponible para cada variedad de uva.
Los métodos de aprendizaje profundo son una alternativa atractiva, pero su rendimiento se ve obstaculizado por conjuntos de datos de fenología escasos, particularmente a nivel de cultivares. En este sentido, Solow y Saisubramanian de la Universidad de Oregon, Estados Unidos (EEUU), proponen un enfoque de modelado híbrido que combina el aprendizaje multitarea con una red neuronal recurrente para parametrizar un modelo biofísico diferenciable.
Al utilizar el aprendizaje multitarea para predecir los parámetros del modelo biofísico, este enfoque permite un aprendizaje compartido entre cultivos, mientras preserva la estructura biológica, mejorando así la robustez y precisión de las predicciones. La evaluación empírica utilizando conjuntos de datos del mundo real y sintéticos demuestra que el método supera significativamente tanto a los modelos biofísicos convencionales como a los enfoques de aprendizaje profundo de referencia en la predicción de etapas fenológicas, así como de otras variables del estado del cultivo, como la resistencia al frío y el rendimiento del trigo.

El enfoque presentado por Solow y Saisubramanian utiliza una red neuronal recurrente que aprende a ajustar diariamente los parámetros del modelo biofísico según las condiciones meteorológicas observadas. Además, incorpora el aprendizaje multi-tarea, lo que permite compartir información entre diferentes variedades de uva sin perder las particularidades biológicas de cada una. De este modo, el sistema puede mejorar su precisión incluso cuando hay pocos datos disponibles para una variedad concreta.
Para validar su propuesta, los autores han utilizado tanto datos reales como simulaciones. Los datos reales proceden del laboratorio Irrigated Agriculture Research and Extension Center de la Universidad Estatal de Washington, donde se han recogido observaciones diarias sobre 32 variedades de uva desde 1988. También se han empleado modelos sintéticos para simular diferentes escenarios climáticos y comprobar la robustez del sistema ante cambios meteorológicos.
Los resultados muestran que el nuevo método reduce el error medio en las predicciones respecto a los modelos tradicionales y a otros enfoques basados en inteligencia artificial. Por ejemplo, en el caso de la predicción fenológica, el error medio se sitúa por debajo de ocho días, frente a los más de 18 días del modelo GDD clásico. Además, el sistema mantiene la coherencia biológica en sus predicciones: no anticipa etapas imposibles (como un retorno a la dormancia tras el brote) y respeta la secuencia natural del desarrollo del cultivo.
El modelo también ha sido probado en otras tareas agrícolas, como la predicción de resistencia al frío en vides y el rendimiento estacional del trigo. En ambos casos, ha superado a los métodos comparativos tanto en precisión como en consistencia biológica.
Introducción
Las tareas estacionales del viñedo, como la fertilización, el riego, la poda y la cosecha, dependen de predicciones precisas de la fenología de la vid. Los principales estados fenológicos de la vid son: brotación, floración y envero. Predicciones inexactas de estos estados pueden llevar a intervenciones mal programadas, lo que resulta en una reducción del rendimiento, la calidad y la salud del viñedo.
Sin embargo, predecir la fenología con precisión es un desafío debido a: 1) la disponibilidad limitada de datos históricos para la calibración por cultivar y las observaciones escasas durante cada temporada de crecimiento, y 2) la necesidad de modelar con precisión las complejas relaciones entre las variables meteorológicas diarias y la fenología. Los enfoques existentes para este problema suelen dividirse en dos categorías: modelos biofísicos mecanicistas y enfoques de aprendizaje profundo basados en datos.

El modelo de Grados-Día de Crecimiento (GDD, por sus siglas en inglés) es un modelo biofísico ampliamente utilizado para la predicción fenológica, que estima las etapas fenológicas en función del calor acumulado diariamente. El modelo GDD se calibra por viñedo utilizando regresión multivariante sobre observaciones históricas de campo. A pesar de la disponibilidad de muchas variables meteorológicas, el modelo GDD sólo usa la temperatura ambiente como entrada, lo que limita su capacidad expresiva. A pesar de varias mejoras propuestas, la precisión de las predicciones fenológicas basadas en GDD sigue siendo relativamente baja, y los viñedos buscan activamente mejoras.
Los métodos de aprendizaje profundo ofrecen una alternativa prometedora debido a su capacidad para modelar relaciones complejas y no lineales entre variables meteorológicas y etapas fenológicas. Un trabajo reciente propuso un modelo de clasificación multitarea para predecir la brotación de la vid, una etapa fenológica clave (Saxena et al. 2023a). Aprovecharon la información compartida entre cultivares para mejorar la precisión, especialmente en cultivares con pocos datos. Sin embargo, este enfoque a menudo produjo predicciones biológicamente inconsistentes, como predecir la brotación seguida de un retorno a la latencia en pocos días, lo cual contradice la progresión unidireccional de las etapas fenológicas. Tales inconsistencias introducen ambigüedad en la interpretación de las predicciones y las vuelven inadecuadas para la toma de decisiones en el campo, especialmente considerando que los viticultores dependen de los modelos fenológicos para realizar pronósticos operativos de mediano plazo (7–14 días).
Una dirección emergente es el modelado híbrido, que combina aprendizaje profundo y modelos biofísicos para abordar sus respectivas limitaciones y mejorar la precisión de las predicciones. Recientemente se ha aplicado para predecir con precisión la fecha de floración en cerezos, aprovechando el aprendizaje profundo para aproximar la función interna de respuesta a la temperatura dentro del modelo GDD. Sin embargo, esta formulación no considera los efectos de variables meteorológicas exógenas, como la irradiación solar y la precipitación, que afectan significativamente el desarrollo fenológico y son particularmente importantes para la predicción fenológica de la vid a lo largo de la temporada.
Para abordar los desafíos en la predicción precisa de la fenología de la vid, los autores de este trabajo proponen un enfoque de modelado híbrido que utiliza un modelo profundo recurrente para predecir los parámetros diarios del modelo GDD, condicionados por variables meteorológicas exógenas. Este enfoque emplea aprendizaje multitarea mediante una incrustación por cultivar para compartir datos de manera eficiente entre cultivares y aumentar la precisión de las predicciones por cultivar.
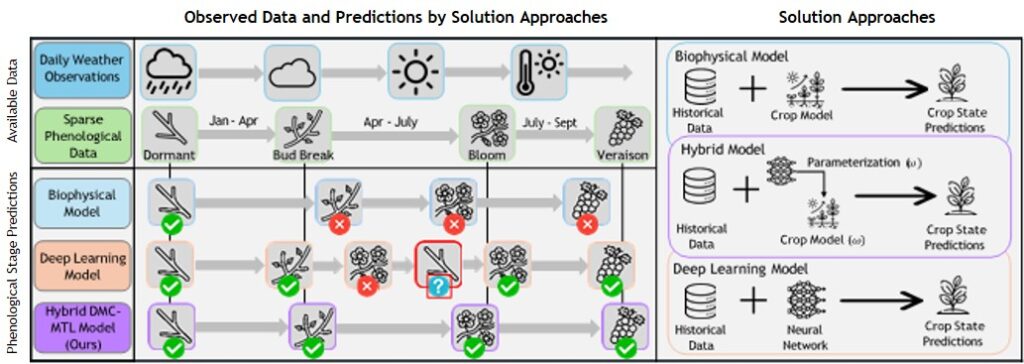
Los autores proporcionan en este estudio una implementación novedosa del modelo GDD en un marco diferenciable, que permite realizar descenso de gradiente sobre los parámetros de la red neuronal con aprendizaje supervisado.
Conclusión y trabajo futuro
Este artículo presenta un novedoso método de aprendizaje profundo que predice los parámetros de modelos biofísicos. Los resultados muestran que aprovechar los beneficios tanto de la arquitectura de redes profundas como de los modelos biofísicos puede superar a ambos métodos por separado.
Se planea implementar el modelo DMC-MTL para fenología en otoño de 2025 en la plataforma ABC (anónima para la presentación). En el futuro, el objetivo es relajar la suposición de un modelo diferenciable mediante un enfoque basado en Aprendizaje por Refuerzo, para explorar otros dominios donde la predicción es crítica para la transferencia de simulación a realidad (por ejemplo, robótica).
Además, también se buscó desarrollar enfoques para la calibración en tiempo real de modelos biofísicos y la cuantificación de la incertidumbre en tareas de predicción del estado de los cultivos.
Ver el estudio completo aquí: https://arxiv.org/abs/2508.03898
Fuente: Universidad de Oregon, Estados Unidos. Título original de la investigación: «Calibración de modelos biofísicos para la predicción de la fenología de la uva a través del aprendizaje multitarea«. Autores: William Solow y Sandhya Saisubramanian, de la Oregon State University.













