Vivero Mercier, licenciatario de la marca ENTAV-INRA® en Argentina, divulgó un informe científico elaborado y publicado por el Instituto Francés de la Vid y el Vino, organismo de referencia a nivel global con el cual el vivero con sede en Agrelo, Luján, Mendoza, mantiene una vinculación institucional desde 2003. El documento revela las últimas novedades en la materia, con datos impactantes sobre cómo la ampelografía molecular y las técnicas genéticas permiten secuenciar el ADN de las plantas y reconocer y diferenciar los cepajes, remontarse hasta su origen y conocer las relaciones entre las variedades de uva. Su autor, el ingeniero agrónomo francés Olivier Yobregat, explica la técnica a partir de 5 preguntas que podrían hacerse los lectores curiosos.
Según el Instituto Francés de la Vid y el Vino, las técnicas modernas para la genotipificación y el análisis de ADN nuclear y de cloroplasto han revolucionado la ampelografía. A través de esta disciplina científica es posible estudiar las relaciones filogenéticas entre especies, diferenciar los grandes grupos dentro de las especies de Vitis vinífera y distinguir entre variedades. También es viable la búsqueda de relaciones entre las variedades de uva y el desarrollo de árboles genealógicos genuinos que han puesto de relieve el papel central de algunos padres importantes en las variedades de uva autóctonas.
Autor: Olivier Yobregat, ingeniero agrónomo, especialista en Material vegetal. Instituto Francés de la Viña y el Vino
1. ¿Qué es la ampelografía molecular?
El término ampelografía proviene del griego «ampelos» que significa vid y de «script» que se relaciona con la descripción. Propuesto por primera vez en 1661 por el doctor Sachs en el libro Ampelographia, este término incluye hoy tanto la descripción como la identificación de las variedades de uva, el estudio de su evolución y las relaciones que existen entre ellas y el conocimiento de su comportamiento hacia el medio ambiente (habilidades culturales y enológicas).

Durante mucho tiempo, la ampelografía se basó únicamente en la observación visual de la vid, en particular en la morfología de las hojas, ramas y racimos. Entre alrededor de un centenar de descriptores codificados para describir los distintos órganos de la vid, la OIV ha mantenido 14 descriptores primarios prioritarios por su buen poder de discriminación entre variedades.
Recientemente, la ampelografía ha adquirido nuevas herramientas como la quimiotaxonomía (estudios de compuestos del metabolismo secundario) o los marcadores bioquímicos (isoenzimas). Sin embargo, estos marcadores han mostrado sus límites, ya que su poder de discriminación resultó insuficiente para su uso a gran escala. La verdadera revolución nació de la incorporación a la ampelografía de técnicas genéticas a partir de los años ‘90. El marcaje molecular y la secuenciación del ADN han hecho posible tener un acceso temprano no solo al fenotipo sino también directamente al genotipo, sin tener que esperar a un desarrollo completo de la planta. El ADN necesario para este tipo de análisis se puede extraer de unas decenas de miligramos de material vegetal y de cualquier órgano o parte de la planta (hojas, bayas, ramas, raíces, etc.).
2. ¿Qué nos enseñó la ampelografía molecular sobre el origen de la vid?
El trabajo llevado a cabo, entre otros, por el equipo de Jean-Pierre Péros del INRA en Montpellier ha permitido resaltar el carácter ancestral de las especies de vid asiáticas, que se dice que ambas son el origen de la especie europea Vitis vinífera y especies americanas. Con respecto a este último, se han producido dos eventos distintos de dispersión de Eurasia a América: un evento en el origen de las especies central y oriental (Vitis labrusca, Vitis riparia, Vitis rupestris y Vitis berlandieri), el otro en el origen de la especie californiana (Vitis californica).
El análisis de los microsatélites también permitió diferenciar entre verdaderas vides silvestres europeas (Vitis vinífera subsp. sylvestris) –también llamados lambrusques– de variedades cultivadas. Dentro de este compartimento cultivado, estos análisis permitieron dividir objetivamente las variedades en cinco grandes grupos geográficos, en función de sus aptitudes.
3. ¿Qué nos enseñó la ampelografía molecular sobre los vínculos de parentesco entre las variedades de uva?
El análisis por microsatélites de las 2.300 variedades de uva tradicionales conservadas en el Domaine de Vassal (INRA de Marseillan) permitió constituir una base de datos completa y única en su tipo. Cuando, en todos los marcadores de una variedad estudiada, podemos encontrar por comparación otras dos variedades de uva que tienen cada una la mitad de los valores determinados, existe una gran probabilidad de que al cruzar estas dos variedades de uva se produzca la tercera. A veces incluso, es posible determinar para una cruza determinada cuál es el padre del que proviene el polen y la madre que dio el fruto. De hecho, es la madre quien proporciona a la futura descendencia toda su «maquinaria metabólica», en particular sus cloroplastos. Estos últimos también contienen ADN sobre el que se han determinado ciertos marcadores genéticos.
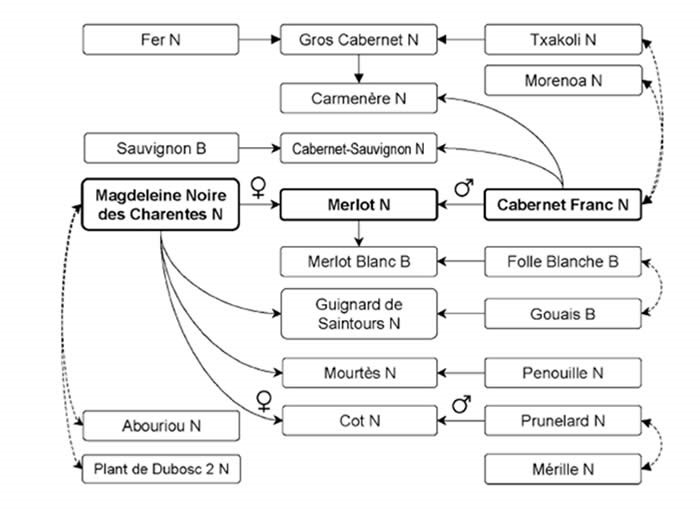
Se ha demostrado así que Gouais B, variedad blanca sin gran interés enológico, fue un importante progenitor de nuestras variedades de uva. Por ejemplo, al cruzar con un Pinot, produjo Chardonnay B, Aligoté B, Auxerrois B, Melon B y Gamay N. Con otros padres a menudo desconocidos, también es originalmente entre otro de Jacquère B, Grolleau N, Colombard B, Riesling B o incluso Saint-Côme B de Aveyron.

Más recientemente, un estudio realizado por Jean-Michel Boursiquot y otros colaboradores, publicado a finales de 2008 en una revista científica australiana, permitió identificar la afinidad de Merlot N. Durante los censos realizados en parcelas viejas de viñas en Bretaña y en Charentes, se ha identificado una variedad de uva original desconocida para las colecciones. Debido a su precocidad, esta variedad fue nombrada «Magdeleine» por ciertos propietarios que la cultivaban en espaldera (la Sainte Madeleine cae el 22 de julio). Luego se le llamó «Magdeleine noire des Charentes» para distinguirlo de otras variedades (especialmente de mesa) ya llamadas Madeleine. Desde entonces, esta variedad de uva también se ha encontrado 3 veces en forma de espaldera en el departamento de Gers, cada vez con el nombre de Madeleine, lo que constituye un hecho bastante destacable. Los análisis genéticos confirmaron que esta variedad, probablemente muy antigua y casi ausente de los textos ampelográficos, era la madre de Merlot N y Cot N (Malbec). También sabíamos que Prunelard N y Malbec estaban emparentados sin conocer previamente el significado de parentesco. Ahora sabemos que Prunelard N es el padre del Malbec.
4. ¿Cuáles son los ejemplos más destacados de identificación de variedades exitosa mediante ampelografía molecular?
En 2009, Jean-Michel Boursiquot y Laurent Audeguin del Plant Material Pole del IFV pudieron identificar un Albarinho B falso durante una misión en Australia. Durante muchos años, los enólogos locales creyeron que estaban cultivando esta variedad originaria de la fachada occidental de la Península Ibérica, pero nuestros colegas rápidamente determinaron que en realidad era Savagnin B. Este resultado se confirmó poco después de su regreso a Francia, gracias al análisis genético de ADN tomado en el sitio y después de la comparación con bases de datos existentes (IFV, INRA). Para la anécdota, la misma historia ya había sucedido en Chile casi veinte años antes y mucho antes del uso de marcadores genéticos. Carmenère N había sido confundido durante mucho tiempo por los viticultores con Merlot N. En la región, estas técnicas han permitido al IFV Sudoeste identificar todas las antiguas variedades mantenidas en los conservatorios regionales, e introducir en las colecciones variedades originales que no figuraban allí, como el Moural N de l’Aveyron, Bouysselet B de Fronton y otras variedades sin denominación hasta la fecha.
5. ¿Puede la ampelografía molecular distinguir entre clones?
Hoy, salvo en algunos casos especiales (ver más abajo), la ampelografía molecular no permite distinguir los clones de una misma variedad de uva, porque hay muy poca diferencia intravarietal a nivel de microsatélites. Sin embargo, los nuevos métodos de secuenciación de ADN de muy alto rendimiento ahora permiten considerar la resecuenciación del genoma de diferentes individuos. Este enfoque se eligió para intentar detectar las diferencias genéticas que pueden existir entre los clones. Durante una tesis realizada por Grégory Carrier en el marco de UMT Géno-Vigne® y supervisada por IFV, Montpellier SupAgro e INRA, se analizaron tres clones de Pinot Noir. Los resultados permitieron destacar el papel preponderante de los elementos móviles (retro-transposones) en el polimorfismo genético detectado entre estos clones. Sobre esta base, un análisis de diversidad preliminar utilizando marcadores SSAP hizo posible obtener perfiles únicos para todos los clones de Pinot Noir en la colección. Sin embargo, aún debe llevarse a cabo una selección de estos marcadores y una validación de su estabilidad y repetibilidad antes de poder prever una identificación fiable de los diferentes clones. Otros trabajos posteriores también permitieron avanzar en la forma de identificar clones. Hoy, por ejemplo, sabemos caracterizar, gracias a un marcador microsatélite específico, la sensibilidad o no de los clones de Syrah al particular declive que afecta a esta variedad de uva. Muy recientemente, los autores han propuesto un nuevo conjunto de marcadores microsatélites capaces de determinar mutantes de color dentro de la misma variedad.
Fuentes: Instituto Francés de la Viña y el Vino y Vivero Mercier Argentina












