En la agricultura de precisión, que utiliza los últimos recursos tecnológicos disponibles y la internet de las cosas, la detección precisa de las enfermedades de la vid es clave para combatir con eficacia sus consecuencias y evitar pérdidas económicas, así como para minimizar el uso de agroquímicos, contribuyendo a la reducción de la huella de carbono y tornando cada vez más sostenible la vitivinicultura.
Sin embargo, las aplicaciones prácticas para la asistencia de los agricultores en este tema son escasas, por lo que un grupo de expertos agrónomos, químicos e informáticos de China, Egipto y Arabia Saudita desarrollaron el software de código abierto y fácil de usar llamado AIGrapeCare. El sistema consiste en el monitoreo en tiempo real de la salud de las vides utilizando técnicas de teledetección de imágenes. Su practicidad y rentabilidad lo convierten en una solución valiosa para la identificación y tratamiento de enfermedades en las plantas. Se emplean técnicas innovadoras para identificar con precisión el estado de salud de las plantas a través del análisis de imágenes.

Por Osama Elsherbiny (de la Escuela de Ingeniería Agrícola, Universidad de Jiangsu, Zhenjiang, China y del Departamento de Ingeniería Agrícola, Facultad de Agricultura, Universidad de Mansoura, Mansoura, Egipto); Ahmed Elaraby (del Departamento de Ciberseguridad, Facultad de Ingeniería y Tecnología de la Información, Buraydah Private Colleges, Buraydah, Arabia Saudita, y del Departamento de Ciencias de la Computación, Facultad de Computación e Información, Universidad del Valle Sur, Qena, Egipto); Mohammad Alahmadi (del Departamento de Ingeniería de Software, Facultad de Ciencias Informáticas e Ingeniería, Universidad de Yeddah, Arabia Saudita); Mosab Hamdan (del Centro de investigación interdisciplinario para sistemas inteligentes y seguros, Universidad Rey Fahd de Petróleo y Minerales, Dhahran, Arabia Saudita) y Jianminn Gao (de la Escuela de Ingeniería Agrícola, Universidad de Jiangsu, Zhenjiang China)
Las uvas son un cultivo de importancia global, con un impacto económico significativo. La existencia de enfermedades en las vides representa una grave amenaza para la seguridad alimentaria mundial, ya que contribuyen significativamente a las pérdidas de cultivos que oscilan entre el 10 y el 30%. Las vides son altamente susceptibles a una amplia variedad de enfermedades fúngicas que pueden reducir los rendimientos. Estas incluyen la mancha foliar (Isariopsis griseola), el moho gris (Botrytis cinerea), el mildiu (Plasmopara viticola), el mildiu polvoriento negro (Guignardia bidwellii). Todas estas enfermedades tienen un efecto adverso en las hojas de las plantas o en el propio cultivo, lo que puede provocar una pérdida moderada a extrema en la producción de uno o ambos.
La salud de las vides puede verse afectada por una variedad de factores, siendo el estrés inducido tanto por elementos bióticos como abióticos. El estrés biótico surge de patógenos vivos como hongos, virus y bacterias, que son los agentes patógenos más prevalentes. En contraste, el estrés abiótico está vinculado a factores no vivos como el clima y las condiciones del suelo. Por ejemplo, la clorosis suele ser un síntoma fisiológico, no una enfermedad en sí misma. Es causada por factores como deficiencias de nutrientes (hierro, nitrógeno, magnesio o zinc), malas condiciones del suelo (drenaje inadecuado, pH alto o suelo compactado) o estrés ambiental (desequilibrio hídrico, daño en las raíces o exposición a contaminantes), todos los cuales provocan el amarillamiento de las hojas.
El método más prevalente para mitigar el estrés biótico en las vides implica la administración de sustancias químicas. Si bien este método ha demostrado ser extremadamente exitoso, también puede tener un impacto perjudicial en el medio ambiente y los ingresos económicos, ya que no siempre es una estrategia rentable.
Se han desarrollado numerosas técnicas de agricultura de precisión en respuesta a los factores mencionados anteriormente en un esfuerzo por maximizar la producción agrícola mientras se disminuye el impacto de factores externos como las plagas y las enfermedades. Como resultado, la industria agrícola hace un uso extensivo de técnicas de detección remota y proximal, así como tecnologías de big data, visión por computadora, robots, técnicas de aprendizaje profundo (DL) y aprendizaje automático (ML) y computadoras de alto rendimiento. Estos métodos tienen utilidad más allá del diagnóstico de enfermedades de las plantas, abarcando la detección de malezas, la evaluación de la calidad del cultivo, la predicción del rendimiento, la identificación de especies, el monitoreo del agua y el suelo, y la gestión del sistema de riego.
Las herramientas informáticas del software AIGrapeCare
La exploración de la arquitectura óptima de aprendizaje profundo implicó la combinación de redes neuronales convolucionales (CNN), redes neuronales recurrentes de memoria a corto y largo plazo (LSTM), redes neuronales profundas (DNN) y redes de transferencia de aprendizaje (incluidas VGG16, VGG19, ResNet50 y ResNet101V2). Se empleó una matriz de co-ocurrencia de niveles de gris (GLCM) para medir las características texturales.
La plataforma de detección de enfermedades de plantas (PDD) creó un conjunto de datos de imágenes reales de hojas de vid de viñedos para mejorar la identificación de enfermedades de las plantas.
Se aplicó una técnica de aumento de datos para abordar el problema de imágenes limitadas.
Posteriormente, el conjunto de datos aumentado se utilizó para entrenar los modelos y mejorar su capacidad para identificar y clasificar con precisión las enfermedades de las plantas en escenarios del mundo real.
Los resultados analizados indicaron que la red profunda combinada CNNRGB-LSTMGLCM, basada en la red preentrenada VGG16 y el aumento de datos, superó las características de la red profunda separada y la versión no aumentada. Su precisión de validación, precisión de clasificación, recuperación y medida F son todas del 96.6%, con una intersección sobre unión del 93.4% y una pérdida de 0.123.
Además, el software desarrollado a través del enfoque propuesto tiene un gran potencial como una herramienta rápida para diagnosticar enfermedades de la vid en menos de un minuto. El marco del estudio muestra potencial para una futura expansión para incluir varios tipos de árboles. Esta capacidad puede ayudar a los agricultores en la detección temprana de enfermedades de árboles, lo que les permite implementar medidas preventivas.
Algoritmos e imágenes digitales
El éxito de los algoritmos de clasificación y las técnicas de imágenes digitales depende de varios factores, como el aprendizaje por transferencia, las características de las entradas de entrenamiento, el aumento de datos y la combinación de múltiples redes profundas entrenadas.
Para resolver problemas de clasificación con un conjunto de datos pequeño, los practicantes de ML pueden recurrir al aprendizaje por transferencia, una técnica que emplea redes previamente entrenadas, típicamente aquellas con arquitecturas profundas. Usando esta estrategia, los pasos de preentrenamiento originales se conservan y se actualizan parcialmente a medida que se introducen nuevos datos en la red.
El punto principal de este método es aprovechar el aprendizaje previo de la red de aprendizaje profundo de modelos de entrenamiento para facilitar el entrenamiento de un nuevo problema de clasificación relacionado que no utilice el mismo espacio de características o distribución. Varios estudios han confirmado que el uso del aprendizaje profundo, particularmente el aprendizaje por transferencia, clasifica eficazmente las enfermedades de las plantas, logrando una precisión de más del 80%. Este enfoque reduce el tiempo computacional y permite el entrenamiento de diversas clases con un número sustancial de instancias, lo que lo hace particularmente adecuado para arquitecturas profundas.
Una variedad de redes preentrenadas bien establecidas, como AlexNet, GoogleNet, ResNet y la familia VGG, están disponibles. Estos modelos DL exhiben variaciones en sus arquitecturas de capas. En el aprendizaje por transferencia, típicamente solo se ajustan los parámetros de la capa final, mientras que el resto de la arquitectura extrae características de las muestras de entrenamiento. Las características de textura basadas en la matriz de co-ocurrencia de niveles de gris (GLCM) también son cruciales para identificar enfermedades en las vides.
Jaisakthi et al. desarrollaron un sistema para detectar enfermedades de la vid, centrándose en la extracción de características de textura como variaciones de color después de la segmentación, para clasificar enfermedades de manera efectiva utilizando algoritmos de máquinas de vectores de soporte (SVM), adaboost y bosque aleatorio (RF). Este método, distinguido por su alta precisión en el diagnóstico de enfermedades como la pudrición y el tizón de las hojas, destaca su valor en la mejora de la gestión de enfermedades agrícolas.
El aumento de datos, considerado durante la fase de entrenamiento en este trabajo, es un factor adicional que puede influir en el rendimiento del modelo de detección. El aumento de datos mediante la aplicación de una secuencia de transformaciones, como el espejo o la rotación de una imagen, aumenta la utilidad y profundidad del conjunto de datos. Además, la fusión de redes profundas se empleó ampliamente para mejorar la calidad y durabilidad del modelo de detección de enfermedades. Anteriormente, este enfoque se había utilizado para identificar fenotipos de plantas. Xiao et al. emplearon una red neuronal convolucional (CNN) con la arquitectura Resnet50 para detectar con éxito diversas enfermedades de la fresa, incluido el moho gris, la corona.
Tradicionalmente, los modelos de DL en agricultura se han basado en datos unimodales, particularmente imágenes de plantas. Sin embargo, las prácticas agrícolas avanzadas han llevado a una tendencia creciente de explotar datos multimodales, que combinan imágenes de plantas con características adicionales como variables GLCM y características pre-entrenadas. Este cambio tiene el potencial de mejorar la precisión y el rendimiento en la estimación de fenotipos de plantas al incorporar diversas fuentes de datos.
Los objetivos de la investigación
El estudio actual se distingue de otras investigaciones en el campo al explorar redes profundas innovadoras, junto con la creación de software independiente diseñado para un modelo de primer nivel. Por lo tanto, los objetivos principales de esta investigación fueron:
1) construir una red profunda híbrida bien organizada para detectar enfermedades en las vides utilizando características de alto nivel extraídas de RGB y GLCM,
2) desarrollar una solución de software independiente y fácil de usar llamada AI GrapeCare para la evaluación y análisis rápido de imágenes digitales relacionadas con la propagación de enfermedades de la vid,
3) explicar los componentes superiores de una red profunda para la detección robusta de infecciones de vid,
4) examinar el comportamiento de las redes profundas en diferentes escenarios que involucran datos aumentados y no aumentados, y
5) comparar el rendimiento de varias redes profundas híbridas que combinan CNN y redes neuronales profundas (DNN) con memoria a corto y largo plazo (LSTM), así como la aplicación de características pre-entrenadas como VGG16, VGG19, ResNet50 y ResNet101V2 durante el entrenamiento. Todos estos procedimientos tienen como objetivo seleccionar el mejor modelo que se pueda recomendar para la agricultura de precisión en el futuro.
Materiales y Métodos
Base de Datos de Imágenes. Para superar las limitaciones de la base de datos PlantVillage para aplicaciones en la vida real, se crearon conjuntos de datos de la plataforma de detección de enfermedades (PDD) para respaldar la planta. Estos conjuntos de datos están accesibles en pdd.jinr.ru, consultados el 10 de diciembre de 2023. La base de datos PDD difiere de PlantVillage ya que abarca diversas condiciones, ángulos y fondos. Este estudio utilizó un conjunto de datos de imágenes de PDD para descubrir enfermedades en las vides, incluyendo específicamente imágenes de la vida real de hojas de vid afectadas por enfermedades. Estas imágenes fueron capturadas directamente de regiones de cultivo de uva.
Como se ilustra en la Figura 1, la base de datos comprende un total de 295 imágenes, que se dividen en 5 categorías distintas de cultivos de uva. Estas clases consisten en 31 imágenes que representan el tizón negro, 49 con clorosis, 73 afectadas por esca, 121 muestras sanas y 22 que muestran el mildiu polvoriento. Todas las imágenes incluidas en la base de datos contienen información relevante sobre tanto los cultivos como las enfermedades presentes en ellos, con un tamaño estandarizado de 256×256 píxeles. Además, recopila imágenes de diversos síntomas de cada enfermedad, lo que nos permite diferenciar enfermedades incluso cuando sus síntomas parecen similares.
Técnicas de Preprocesamiento de Imágenes. Para garantizar un entrenamiento efectivo del modelo y minimizar cualquier defecto que pueda surgir durante el proceso de imagen, es necesario realizar preprocesamiento de imágenes RGB antes de proceder con el análisis de datos. La fase de preprocesamiento implica varias etapas, incluida la segmentación para eliminar cualquier elemento de fondo, el aumento de datos para aumentar el tamaño del conjunto de datos de entrenamiento y la transformación de características para normalizar y estandarizar las características de las imágenes.
Primero, la separación del fondo es esencial para aislar la planta de vid y eliminar cualquier elemento ajeno de las imágenes. Se utilizó un proceso de segmentación llamado técnica de umbral, que implica convertir la imagen a escala de grises y producir una imagen binaria.
La imagen tiene dos posibles valores de píxel: un valor igual a 1, que representa los píxeles de la vid, y un valor igual a 0, que significa píxeles no relacionados con la vid que pueden ser excluidos. La imagen es binaria, con cada píxel representado por un solo bit. Una vez separados los píxeles de la vid del fondo, se obtuvo un conjunto de características de color potenciales para un análisis adicional.
Segundo, el aumento de datos es un paso crucial para mejorar el proceso de aprendizaje de una red profunda y para permitirle identificar objetos en imágenes capturadas en diversas condiciones del mundo real. En este trabajo, se realizaron varias técnicas de aumento para aumentar la confianza en el proceso de categorización. Estas incluyeron el uso de la imagen original, aplicar un rango de zoom de 0.3, rotar la imagen hasta 90 grados, voltearla horizontalmente y desplazar su ancho en un rango de 0.1 y su altura en un rango de 0.2.
Tercero, para abordar las variaciones en las magnitudes de las características, se aplica normalización a cada característica individualmente. El cálculo de normalización se deriva dividiendo el rango entre los valores máximos y mínimos de las características por el valor mínimo de datos de imagen.
Características de Textura Derivadas de la Matriz de Co-ocurrencia de Niveles de Gris. El uso de la matriz de co-ocurrencia de niveles de gris (GLCM) para detectar y diagnosticar enfermedades de plantas ha sido ampliamente estudiado en varios trabajos de investigación. Yogeshwari y Thailambal emplearon una GLCM como método de extracción de características en su marco para detectar enfermedades de hojas de plantas, capturando exitosamente detalles de textura de las imágenes. Sari et al. emplearon características de textura y un modelo SVM para la clasificación de enfermedades de hojas de chile.
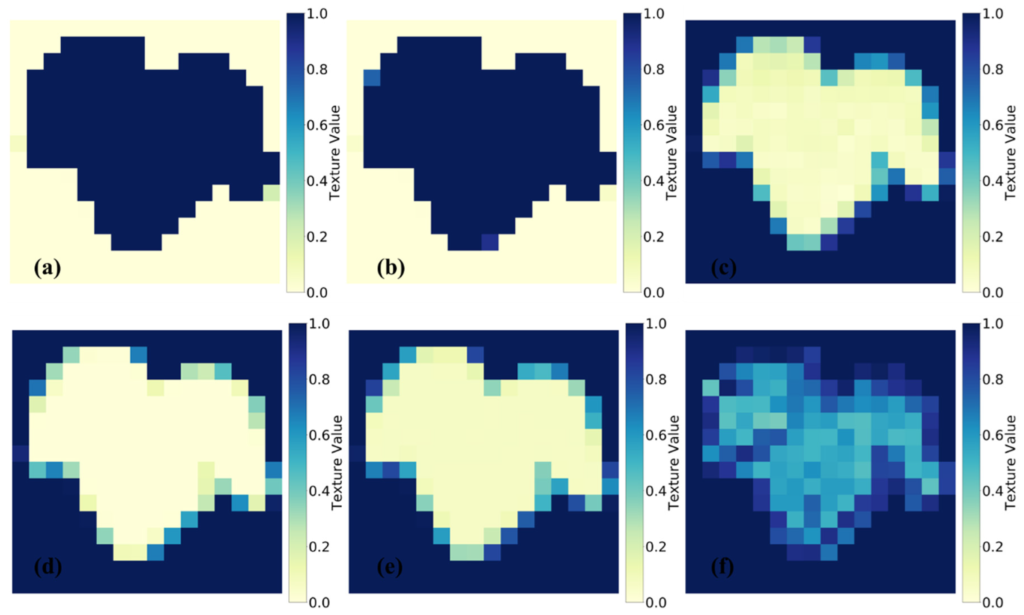
La matriz de co-ocurrencia es una técnica estadística empleada para el análisis de textura en una imagen en escala de grises. Al examinar la correlación en escala de grises entre dos píxeles en el espacio de imagen separados por una distancia específica, la GLCM extrae las características de textura del dosel a través de características de probabilidad de manera aleatoria.
Este estudio involucró seis versiones diferentes de la GLCM, a saber, contraste, disimilitud, homogeneidad, momento angular segundo (ASM), energía y correlación, como técnicas de extracción de características. La profundidad y textura de una imagen se indican por su contraste, mientras que la disimilitud calcula la separación entre pares de píxeles dentro de un área especificada. La homogeneidad evalúa qué tan cercana es la distribución de elementos en la GLCM, mientras que ASM detecta la rugosidad de la distribución y textura de la imagen.
La energía es una medida de la uniformidad de la textura, y la correlación determina el grado de correlación presente en la imagen en escala de grises local.
Como se muestra en la Figura 2, este trabajo utiliza la metodología de GLCM para extraer información de textura de hojas de vid segmentadas. El algoritmo de GLCM calcula características de textura para cada región de píxeles que desempeña un papel crucial en los análisis posteriores. Las siguientes fórmulas, según lo descrito en Hall-Beyer, proporcionan una explicación detallada de las variables específicas derivadas del enfoque de GLCM basado en RGB.

Resumen del enfoque sugerido
Múltiples redes profundas, que incluyen arquitecturas CNN, DNN, CNN-LSTM y DNN-LSTM, fueron sometidas a entrenamiento utilizando una amplia gama de datos multimodales, que incluyeron variables GLCM, características pre-entrenadas y datos de imagen. Las características pre-entrenadas fueron obtenidas de VGG16, VGG19, ResNet50 y ResNet101V2.
Generalmente, CNN y DNN fueron entrenadas utilizando datos de imagen, mientras que LSTM utilizó factores GLCM para la identificación de enfermedades en cultivos de vid, por ejemplo, tizón negro, clorosis, esca, mildiu polvoriento y saludable.
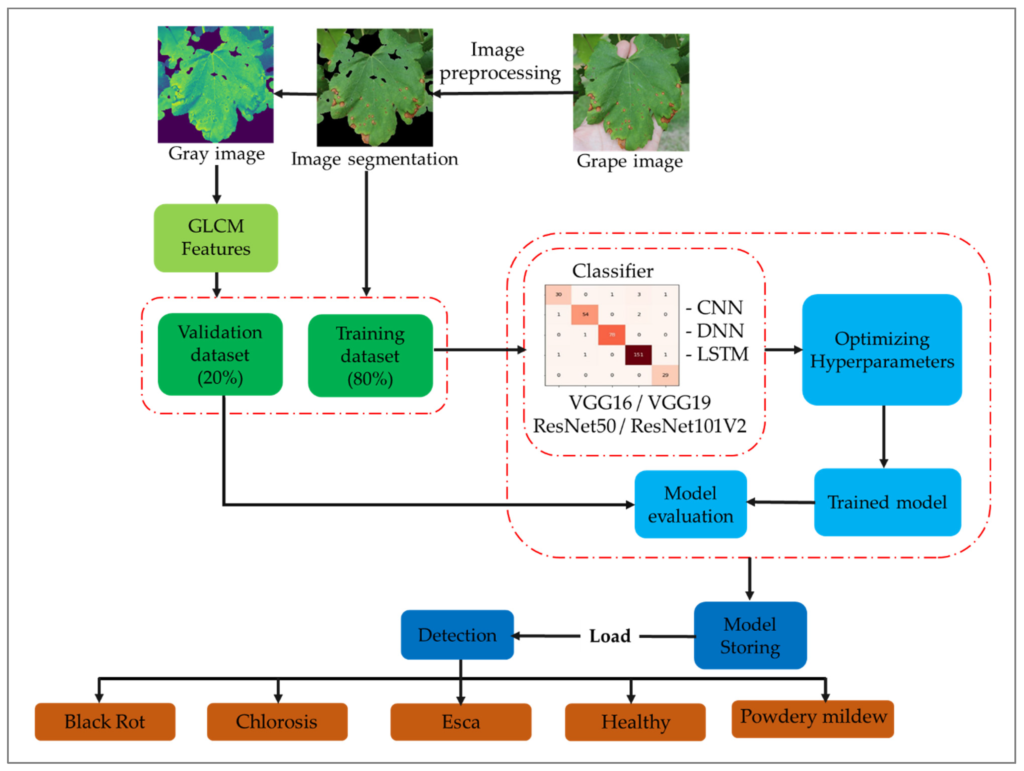
Los pasos secuenciales del marco propuesto se muestran en la Figura 3: (a) realizar tareas de preprocesamiento de imágenes como normalización, segmentación y aumento; (b) convertir las imágenes a escala de grises para extraer características GLCM; (c) dividir el conjunto de datos y entrenar redes neuronales profundas; (d) entrenar los modelos bajo diferentes escenarios, incluyendo CNN, DNN, CNN-LSTM y DNN-LSTM; (e) incorporar características pre-entrenadas extraídas de VGG16, VGG19, ResNet50 y ResNet101V2 durante el proceso de entrenamiento; (f) optimizar los hiperparámetros para seleccionar la mejor configuración durante el entrenamiento; (g) analizar el rendimiento del modelo en nuevas muestras; (h) evaluar el rendimiento general del modelo de clasificación; (i) guardar el modelo avanzado para uso futuro; y (j) cargar el modelo de red avanzado para apoyar a los gestores de enfermedades de la vid en la toma de decisiones efectivas.
AI GrapeCare, un software para el análisis de la salud de la uva
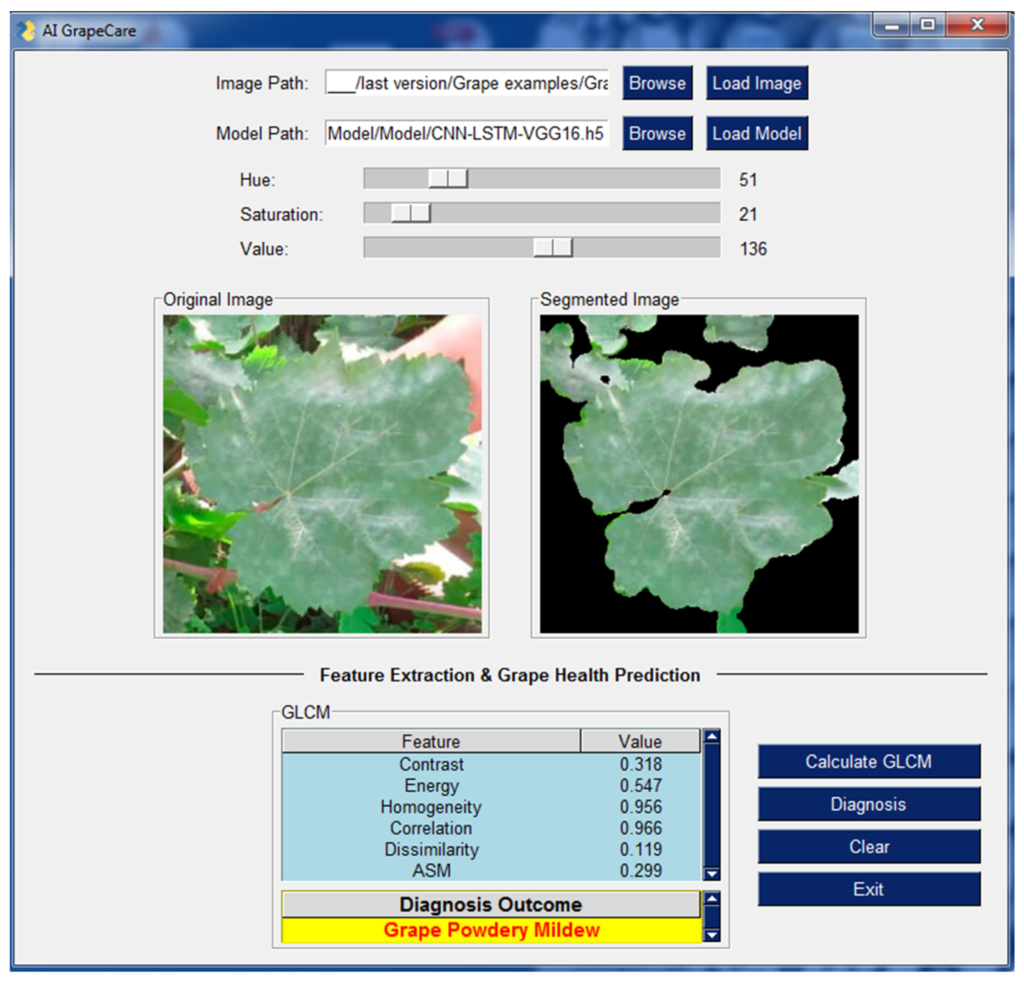
Como se muestra en la Figura 10, la interfaz del software es fácil de usar y no requiere habilidades de programación. El software es capaz de cargar y analizar imágenes digitales con varias extensiones, como PNG, JPG, JPEG y TIFF. Además, admite la carga de un modelo híbrido profundo superior llamado CNNimg-LSTMGLCM-VGG16. El software incluye procedimientos de preprocesamiento de imágenes, como la segmentación de imágenes y la compresión.
Durante la segmentación de imágenes, el software AI GrapeCare puede ayudar a los usuarios a seleccionar los parámetros óptimos mostrando visualizaciones de las imágenes segmentadas que genera. Las características útiles (características texturales) pueden extraerse y mostrarse en una GUI basada en tablas, listas para ser utilizadas como entradas para que el modelo evalúe el estado del crecimiento de la vid. El tiempo de diagnóstico del software AI GrapeCare para evaluar las condiciones de salud de las uvas suele ser de solo unos segundos y nunca supera 1 minuto. El rendimiento del modelo del software se registró en un 96.6%, con una pérdida de 0.123.
Este estudio presenta un enfoque híbrido novedoso impulsado por una interfaz gráfica de usuario (GUI) para el monitoreo en tiempo real de la salud de las vides utilizando técnicas de teledetección basadas en RGB. Su practicidad y rentabilidad lo convierten en una solución valiosa para la detección de enfermedades en las plantas. Se emplean técnicas de modelado innovadoras para identificar con precisión el estado de salud de las plantas a través del análisis de imágenes.
El modelo avanzado desarrollado demuestra un rendimiento excepcional en la clasificación de enfermedades y la reducción de pérdidas, lo que indica su efectividad. La integración de sistemas basados en UAV, que utilizan teledetección de alta resolución espacial, puede mejorar el monitoreo de áreas agrícolas a gran escala y mejorar significativamente la gestión de enfermedades. Además, la implementación de sistemas de transmisión de datos RGB basados en IoT permite un enfoque no destructivo y eficiente para monitorear la salud de las vides y diagnosticar enfermedades.
Esta tecnología facilita la recopilación oportuna de datos, minimiza la interrupción de las plantas y acelera el procesamiento de datos de teledetección, lo que conduce a una toma de decisiones más rápida y fundamentada en la agricultura de precisión. Las valiosas ideas de este estudio contribuyen a los esfuerzos continuos para implementar prácticas agrícolas efectivas. Al aprovechar los avances en imágenes digitales y aprendizaje profundo, esta investigación contribuye al desarrollo de soluciones automatizadas que mejoran la detección de enfermedades y aumentan la eficiencia en la gestión agrícola.
Finalmente, aunque esta investigación puede detectar la clorosis en las vides, es importante tener en cuenta que la clorosis puede resultar de varios factores, como se menciona en la literatura. Por lo tanto, los estudios futuros deberían centrarse en desarrollar un modelo capaz de reconocer eficazmente la clorosis y comprender sus causas subyacentes como síntoma. Esto es crucial debido a la naturaleza multifacética de la manifestación, lo que plantea desafíos significativos en la identificación precisa. Al concentrarse en este aspecto, los investigadores pueden abordar mejor las complejidades involucradas en la especificación de la clorosis.
Además, nuestra metodología desarrollada es competente para clasificar con precisión enfermedades individuales en las hojas. Sin embargo, hay una necesidad imperativa de avanzar en técnicas de diagnóstico que puedan distinguir y descubrir simultáneamente varios patógenos que exhiben síntomas similares en la misma hoja. La implementación de algoritmos de detección de objetos con dos o más clases puede ser instrumental en estas mejoras en el examen, proporcionando una comprensión en capas y comprensiva de la patología de la vid.
Conclusiones
La vid, un cultivo de frutas de importancia global, a menudo se ve afectada por cuatro enfermedades prevalentes: esca, clorosis, mildiu polvoriento y podredumbre negra. El diagnóstico oportuno y preciso juega un papel fundamental en contener la propagación de enfermedades y minimizar las pérdidas de producción en la vid. El progreso en el aprendizaje profundo ha abierto el camino para algoritmos de diagnóstico innovadores en el campo de la identificación de enfermedades de las plantas, desbloqueando nuevas posibilidades y vías para una detección precisa y eficiente.
Este documento presenta un enfoque híbrido innovador, utilizando un software independiente llamado AI Grapecare, para procesar y analizar imágenes RGB para identificar enfermedades de la vid. El marco propuesto integra varios redes de aprendizaje profundo, incluyendo una red neuronal convolucional (CNN), una memoria a largo plazo a corto plazo (LSTM) y una red neuronal profunda (DNN). También emplea características multimodales como VGG16, VGG19, ResNet50 y ResNet101V2, junto con características texturales basadas en la matriz de co-ocurrencia de niveles de gris (GLCM).
Los modelos híbridos, como CNNimg-VGG16, CNNimg-LSTMGLCM-VGG16, DNNimg-VGG16 y DNNimg-LSTMGLCM-VGG16, se entrenaron con el 80% de los datos, mientras que el 20% restante se reservó para la evaluación. Esta metodología permitió una evaluación exhaustiva del rendimiento, lo que permitió la selección del modelo más efectivo. Basándose en los resultados experimentales, la red híbrida de CNNimg-LSTMGLCM-VGG16 exhibió una capacidad predictiva notable como extractor de características y clasificador para el diagnóstico de enfermedades de la vid. Al incorporar sus características extraídas, el sistema propuesto logró un rendimiento de clasificación impresionante, con precisión, recall y F-measure alcanzando el 96.6% y una intersección sobre la unión del 93.4%. La precisión de validación del sistema fue del 96.6% con una pérdida de 0.123.
El enfoque propuesto basado en AI GrapeCare demostró la superioridad de la arquitectura del modelo elegido, destacando su eficacia en la clasificación de hojas de enfermedades de la vid con un tiempo de procesamiento y una participación laboral mínimos. Se espera que AI GrapeCare mejore la eficiencia de la fenotipificación en la agricultura de precisión mediante el análisis de la dinámica de salud de las frutas, lo que lleva a un cultivo consistente de cultivos de alta calidad.
Fuente: Artículo “Rapid Grapevine Health Diagnosis Based on Digital Imaging and Deep Learning”, publicado originalmente en la revista Plants en enero de 2024 (doi: 10.3390/plants13010135)