A través de su evolución, las cepas enológicas de S. cerevisiae han adquirido una serie de características genéticas y genómicas que les han permitido predominar en este ambiente, haciéndolas más tolerantes a factores de estrés tanto generales como específicos de la fermentación. El conocimiento de estas adaptaciones es muy relevante para el desarrollo, como cultivos iniciadores, de nuevas cepas enológicas de S. cerevisiae con características alineadas con las demandas actuales del mercado. En este artículo se revisarán las características genéticas que permiten a S. cerevisiae imponerse en este ecosistema y finalizar la fermentación.

El mosto de uva, como ecosistema en el que ha de producirse la fermentación alcohólica, es un medio bastante agresivo para los microorganismos. Se caracteriza por una alta concentración de azúcares (que generan estrés osmótico), bajo pH, y presencia limitada de nitrógeno y vitaminas. Todo ello, junto con el uso del sulfitado, hace que sólo aquellos microorganismos mejor adaptados a estas condiciones sean los que se impongan durante la fermentación.
Entre la amplia biodiversidad de microorganismos presente en la superficie de la uva antes del prensado, Saccharomyces cerevisiae es la levadura con mecanismos mejor adaptados para competir e imponerse durante la fermentación. Estos mecanismos han sido adquiridos y optimizados a lo largo del proceso de domesticación llevado a cabo de forma involuntaria por el ser humano.
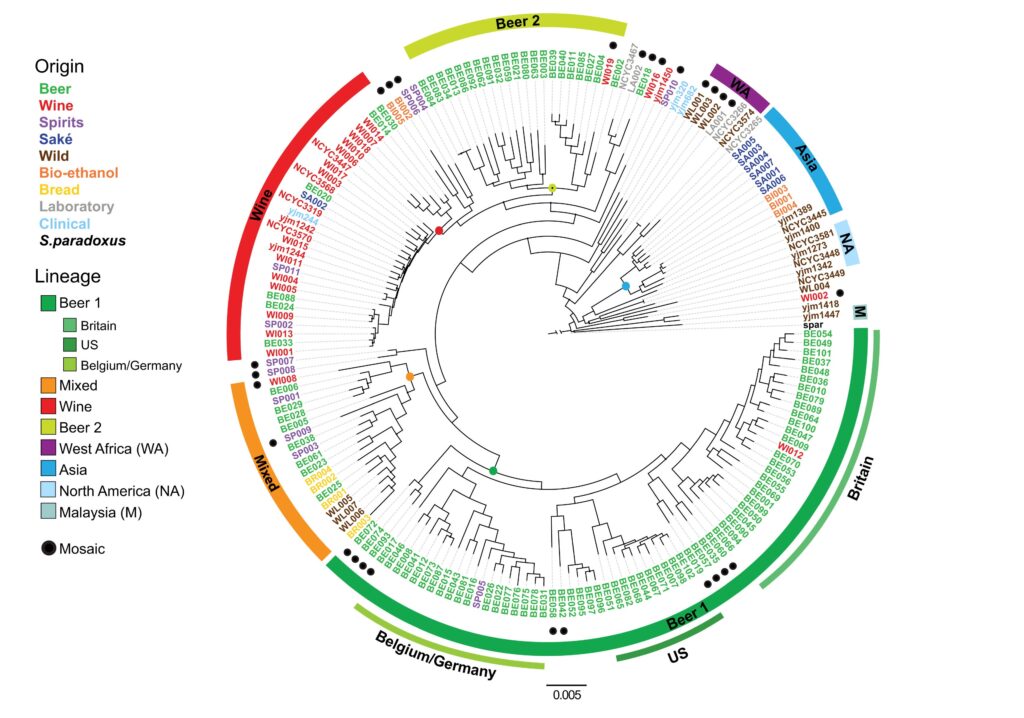
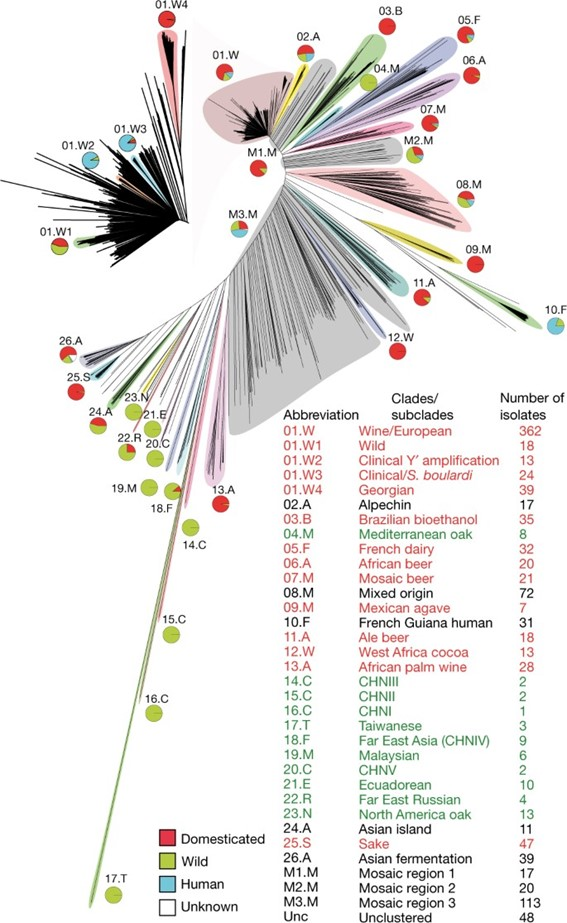
Estas adaptaciones propias de los entornos enológicos no están presentes de la misma manera en todas las cepas de S. cerevisiae. El clado (grupo de microorganismos que descienden de un ancestro común y sus descendientes directos, tanto vivos como extintos) formado por las cepas vínicas representa sólo una pequeña parte de toda la biodiversidad de cepas que se han adaptado a ambientes humanos y procesos industriales como pueden ser la elaboración de cerveza o sake y las cepas aisladas de medios naturales (Fig. 1). Aun así, forma un conjunto de cepas bien definido por sus características genéticas.
Algunas de las adaptaciones para soportar el estrés osmótico son la sobreproducción de glicerol y una cascada de MAP kinasas específica, la ruta HOG. Estas adaptaciones no son las únicas necesarias para soportar el estrés osmótico, sino que forman parte de mecanismos de control más amplios, al igual que la tolerancia a altas concentraciones de etanol o temperaturas extremas.
Para responder de forma coordinada a estas fuentes de estrés, en S. cerevisiae se han descrito dos mecanismos de respuesta generales, el ESR (Environmental Stress Response) y el FSR (Fermentation Stress Response). Se trata de cascadas de respuestas al estrés que a su vez comparten hasta un 20% de los genes implicados. Estas adaptaciones y mecanismos de respuesta no son exclusivos de las cepas vínicas de S. cerevisiae, tampoco de la especie o el género, pero sí se trata de las cepas más eficaces a la hora de responder a estos estímulos.
Esta eficacia es la que permite a S. cerevisiae imponerse en un ambiente en el que, en un primer momento, se trata de una especie minoritaria. La mayoría de microbiota presente en las uvas no sobrevivirá a las primeras horas de fermentación. Algunos géneros de ascomicetos como Hanseniaspora, Torulaspora, Metschnikowia, Starmerella, Candida o Pichia estarán presentes durante un mayor número de horas y contribuirán a la fermentación junto a S. cerevisiae. Finalmente, y gracias a las adaptaciones descritas, S. cerevisiae se impondrá y finalizará la fermentación.
En este artículo se revisarán las características genéticas que permiten a S. cerevisiae imponerse en este ecosistema y finalizar la fermentación.
Evolución del genoma de S. cerevisiae
Durante su evolución, el genoma de S. cerevisiae se ha modificado constantemente. Entre estas modificaciones, se destaca la duplicación del genoma (WGD por sus siglas en inglés). Este evento, que consiste en que todo el genoma de un ancestro común de Saccharomyces y otros géneros de levaduras se duplicó para dar lugar a una célula tetraploide, ocurrió aproximadamente hace 100 millones de años. Tras la duplicación se produjo una pérdida de muchos de los genes duplicados y una reordenación cromosómica, conservando hasta un 13% de los genes duplicados. Entre los efectos más relevantes de esta duplicación se observa un flujo glucolítico aumentado gracias a la conservación de copias duplicadas de genes relacionados con la glicolisis. Un ejemplo de esta duplicación son los genes ADH1 y ADH2, los cuales contribuyen a un uso más eficiente de la glucosa como fuente de carbono durante la fermentación y la utilización del etanol como fuente de carbono (crecimiento diaúxico).
La duplicación del genoma también permitió la aparición de la estrategia MAC (make-accumulate-consume) la cual implica el aumento del flujo glicolítico, el efecto Crabtree y el crecimiento diaúxico. Esta estrategia permite a S. cerevisiae utilizar rápidamente la glucosa durante la fermentación produciendo etanol (make), mantener el metabolismo fermentativo para producir etanol independientemente de la presencia de oxígeno en el medio (acumulate) y una posterior utilización del etanol como fuente de carbono (consume). Si bien en condiciones de vinificación esta última etapa no ocurre habitualmente (salvo en los vinos de crianza biológica) debido a la pérdida de viabilidad y la ausencia de oxígeno, que es necesario para metabolizar el etanol.
Detoxificación del sulfito
El género Saccharomyces presenta diversos mecanismos para mitigar el efecto del sulfito, los cuales incluyen un incremento en los niveles de acetaldehído, que se combina con sulfito, la regulación de la ruta metabólica para la asimilación de sulfito y la detoxificación de sulfito expulsándolo del medio celular. Este último mecanismo es llevado a cabo por una transportador de membrana codificado por el gen SSU1.
Cabe destacar que las cepas vínicas son más resistentes al dióxido de azufre que las cepas de otros entornos. Esta característica diferenciadora se puede atribuir a la presión selectiva ejercida por el ser humano durante el proceso de domesticación de la levadura para la producción de vino.
Hasta la fecha se han descrito tres reordenaciones cromosómicas diferentes que mejoran la detoxificación del sulfito en cepas enológicas de S. cerevisiae. Cada una de ellas da lugar a la sobreexpresión del gen SSU1 mediante la modificación de su promotor. La translocación recíproca de SSU1 entre los cromosomas VIII y XVI genera un alelo SSU-R con mayor nivel de expresión que el gen nativo debido al intercambio del promotor con el gen constitutivo ECM34. La translocación entre los cromosomas XV y XVI (XVtXVI) posiciona el gen SSU1 bajo el control del promotor del gen ADH1, el cual está expresado de forma constitutiva. Por último, la tercera reordenación cromosómica que confiere resistencia al sulfito es una inversión cromosómica de 38,5 kb en el cromosoma XVI. Esta inversión confiere una tolerancia al sulfito similar a las reordenaciones mencionadas anteriormente.
Estas tres reordenaciones han ocurrido de manera independiente y tienen un resultado final similar, aunque con algunas características diferenciadoras. La translocación VIIItXVI es la más abundante en las poblaciones estudiadas, y la translocación XVtXVI es la más eficiente en términos de resistencia al sulfito. Recientemente se ha identificado una reordenación cromosómica en Saccharomyces uvarum independiente de las observadas en S. cerevisiae. Las reordenaciones previamente descritas y el ejemplo identificado en S. uvarum son ejemplo de cómo la presión selectiva ejercida por el ser humano genera eventos de evolución convergente.
Introgresiones en S. cerevisiae
Tras la secuenciación de 1011 genomas de S. cerevisiae se identificaron 913 ORF (Open Reading Frame) procedentes de Saccharomyces paradoxus en el pangenoma de S. cerevisiae. De las 1.011 cepas secuenciadas, todas tenían al menos una ORF procedente de S. paradoxus, siendo la media de ORF por genoma de 26. La presencia de estas introgresiones demuestra una trasferencia de genes mediante hibridación interespecífica entre estas dos especies y que esta transferencia no es un evento singular, si no que está presente en mayor o menor medida en todas las cepas estudiadas.
Las introgresiones de S. paradoxus en el pangenoma de S. cerevisiae no son exclusivas de esta especie. Se han identificado diversas contribuciones al pangenoma de S. cerevisiae por otras especies más alejadas filogenéticamente mediante transferencia horizontal.
El primer ejemplo detectado en una cepa vínica es el de EC1118, donde se detectaron tres regiones denominadas A, B y C procedentes de las especies Zygosaccharomyces bailii, Torulaspora microellipsoides y Torulaspora delbrueckii. Algunos de estos eventos de transferencia horizontal se han relacionado con la adquisición de adaptaciones a un entorno fermentativo.
La adición al pangenoma de S. cerevisiae de una dehidrorootato deshidrogenasa de origen bacteriano supone a nivel filogenético una de las adaptaciones más relevantes a entornos fermentativos. Aunque los organismos eucariotas poseen esta enzima, necesaria para la síntesis de la pirimidina, su actividad es dependiente de enzimas de la cadena respiratoria. La adquisición del gen de origen bacteriano por algunos integrantes de la familia Saccharomycetaceae les ha permitido la síntesis de pirimidina en ausencia de oxígeno utilizando el fumarato como aceptor de electrones. Este evento de transferencia horizontal supuso una clara ventaja a la hora de imponerse en medios anaerobios como el que se produce durante la fermentación del mosto, y se ha relacionado con la estrategia MAC.
Plásmido de 2 micrómetros
Uno de los elementos extracromosómicos más comunes en procariotas son los plásmidos. Sin embargo, estos fragmentos circulares de DNA también pueden aparecer en eucariotas. En concreto, en el núcleo de S. cerevisiae encontramos frecuentemente el plásmido de 2 micrómetros. Se encuentra presente en muchas levaduras vínicas portadoras en un número de copias variable entre 1 y 210, y tiene un tamaño de 6,3 kb. Considerado un elemento de naturaleza “egoísta” codifica únicamente para cuatro proteínas (Rep1, Rep2, Raf1 y Flp) relacionadas con la segregación del plásmido entre las células hijas, mantenimiento del número de copias en la célula y estabilidad de la molécula. Debido precisamente a su gran estabilidad y alto número de copias, se emplea ya desde los años 80 como vector para la expresión de genes de interés. Se han identificado tres tipos diferentes de este plásmido, el A, el C y el B, siendo este último una versión híbrida entre las otras dos. El tipo A es el que tiene mayor representación en cepas enológicas de S. cerevisiae.
Genoma mitocondrial
En cada célula haploide de S. cerevisiae existen aproximadamente de 50 a 200 copias de DNA mitocondrial, lo cual corresponde al 15% del DNA total en la célula. Está compuesto en su mayoría por fragmentos lineales de 75 a 150 kb, aunque también encontramos un pequeño número de moléculas circulares. Una característica reseñable es que en S. cerevisiae el DNA mitocondrial posee un bajo contenido general de GC, así como regiones intergénicas extensas compuestas principalmente por adenosina y timina que conforma el 62% del genoma mitocondrial.
A pesar de su importancia para diversos procesos celulares, mutaciones que afectan a la fosforilación oxidativa (fenotipo petite) e incluso la pérdida total del genoma mitocondrial (fenotipo rho0), no resultan letales para S. cerevisiae siempre que existan fuentes de carbono fermentables (Chen y Clark-Walker 2000).
Sin embargo, algunos estudios afirman que estas deficiencias en el DNA mitocondrial comprometen la supervivencia de Saccharomyces a altas y bajas temperaturas o afectan a la tolerancia al etanol. De hecho, se ha observado que la transferencia del genoma mitocondrial de levaduras provenientes de velo de flor de vinos de Jerez a levaduras rho– es capaz de conferir resistencia a etanol. Adicionalmente, la herencia mitocondrial parece tener un gran impacto en la resistencia a estrés oxidativo y al proceso de deshidratación realizado en la industria durante la producción de cultivos iniciadores.
Debido a estas implicaciones, es clara la importancia del genoma mitocondrial para la industria vitivinícola. De hecho, hace unos años se desarrolló un innovador método basado en los patrones de restricción del DNA mitocondrial que permite diferenciar entre cepas de S. cerevisiae, distinguiendo así la cepa inoculada de las cepas salvajes presentes en el mosto. Esta técnica se ha propuesto como un control de que el proceso de fermentación está realmente dirigido por la cepa inoculada. Siguiendo esta línea, se ha descrito un método de PCR basado en las variaciones en el número y posición de intrones en el gen mitocondrial COX1 ente distintas cepas de S. cerevisiae con la ventaja de que no requiere la purificación de DNA genómico.
Ciclo de vida de S. cerevisiae
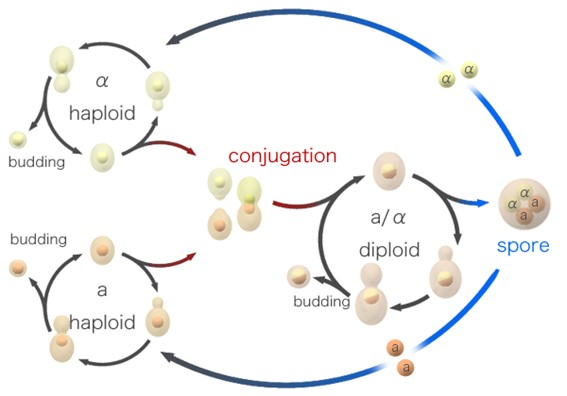
S. cerevisiae puede experimentar tanto reproducción sexual como asexual, permitiendo su adaptación a distintas condiciones ambientales. Si éstas son óptimas, se multiplicará por gemación mediante una mitosis asimétrica dando lugar a una protuberancia que finalmente se convertirá en la célula hija (Fig. 2). Este proceso pueden sufrirlo tanto células haploides como diploides. Prácticamente todas las células enológicas son diploides debido posiblemente a su estabilidad genética, tolerancia al estrés y capacidad de adaptación.
Sin embargo, si las condiciones no son favorables, las células diploides pueden llevar a cabo la meiosis, para generar un asca con cuatro ascosporas haploides sexualmente diferentes, dos de tipo MATa y dos MATα. Éstas pueden combinarse a su vez para formar una célula diploide MATa/α (Fig. 2). El tipo sexual viene determinado por el locus MAT que se encuentra en el cromosoma III de S. cerevisiae. Cada uno de los alelos MAT expresa proteínas diferentes que, a través de una serie de señales de activación o represión transcripcional, van a regular el comportamiento sexual de las células.
Además, en S. cerevisiae diferenciamos entre cepas homotálicas y heterotálicas. Las cepas homotálicas son las más comunes en la naturaleza y pueden llevar a cabo un cambio sexual. A partir del segundo ciclo de división, una célula haploide que haya llevado a cabo la mitosis puede cambiar su condición sexual favoreciendo así la formación de diploides homocigotos. Durante este proceso de cambio, el alelo para MAT es reemplazado por el alelo opuesto gracias a la acción de la endonucleasa HO. En cada extremo del cromosoma III se encuentra el locus HMRa y el HMLα, que contienen la información para generar cada cassette MAT, pero permanecen silenciados. De esta forma, la endonucleasa HO genera una ruptura de doble hebra en el locus MAT que desencadena el reemplazo por el alelo contrario empleando la información genética de HMRa o HMLα. Este proceso está en consonancia con la propuesta de “renovación del genoma” en la que las cepas vínicas pueden sufrir cambios que las transformen de una población heterocigota a una homocigota eliminando alelos deletéreos o letales.
En cuanto a las cepas heterotálicas, pueden mantenerse como haploides de forma estable debido a la pérdida de la funcionalidad del gen HO, lo que las convierte en una herramienta muy útil para la investigación.
Siguiendo esta línea, se ha observado que la generación de híbridos entre cepas de S. cerevisiae puede dar lugar a fenotipos con mayor capacidad de fermentación y producción de metabolitos secundarios (Bonciani et al. 2016). Asimismo, algunos estudios muestran que la hibridación entre especies de Saccharomyces puede generar nuevas cepas, algunas de las cuales se han seleccionado por su impacto en el resultado final del vino. Se han descrito híbridos que aportan una serie de características relevantes para la industria como la reducción en la concentración de L-malato y sulfitos o diferencias en el perfil aromático, concentración de glicerol y etanol respecto a sus progenitores.
Fenotipo killer
El fenotipo killer en S. cerevisiae consiste en la producción de una serie de toxinas de naturaleza proteica, que pueden matar o inhibir el crecimiento de otras cepas de levadura y hongos filamentosos susceptibles, sin perjudicar a la levadura que las produce. Aunque se ha detectado su producción por otras especies de levaduras y géneros de hongos, fueron descubiertas inicialmente en S. cerevisiae, en la cual se conocen específicamente las toxinas K1, K2, K28, Klus, KHR1 y KHS1. A excepción de KHR1 y KHS1, que se han identificado en el genoma nuclear, el resto están codificadas por RNA de doble cadena asociados a micovirus de las familias Totiviridae y Partitiviridae.
La transmisión de estos virus es tanto vertical (de madre a hija durante la división celular), como horizontal (mediante la fusión de células), como en la hibridación sexual. Sin embargo, no se ha identificado una vía de transmisión extracelular. En el citoplasma de la célula se encuentran las partículas virales cuya cápside encierra dos tipos de genoma. El más extenso, llamado genoma “L”, codifica para las proteínas de la cápside y la maquinaria replicativa. El genoma “M” codifica para la toxina. Estas toxinas actúan en la célula a través de distintos mecanismos. La mayoría se une a proteínas de la pared celular del microorganismo objetivo, provocando la formación de poros en la membrana plasmática.
Sin embargo, la toxina K28 es internalizada por endocitosis y posteriormente llega al núcleo donde bloquea el ciclo celular. En el contexto del vino, las toxinas producidas por distintas levaduras ofrecen una oportunidad para el control de crecimiento microbiano. De hecho, se han propuesto cepas de S. cerevisiae como agentes de control de otros organismos durante la fermentación, con impacto sobre el perfil aromático del vino. Hay que destacar que en fermentaciones espontáneas del suroeste de España se han encontrado levaduras que producen principalmente K2 y de forma minoritaria Klus. Adicionalmente, se ha observado que ambas toxinas son muy activas a valores de pH relevantes para la elaboración del vino.
Además de los virus de las toxinas killer, existen otros elementos que se heredan de forma no mendeliana en S. cerevisiae, como los elementos de tipo prion. Se trata de proteínas que pueden cambiar a una conformación alternativa que se propaga por interacción con las proteínas en la conformación principal, afectando a su función. Se ha propuesto que algunos elementos de tipo prion podrían ser relevantes en la ecología de las levaduras enológicas, pero estos resultados son controvertidos.
Otros elementos que constituyen una fuente importante de variabilidad genética son los retrotransposones, que se transcriben a RNA y a continuación se retrotranscriben a DNA para insertarse en nuevas ubicaciones del genoma. En S. cerevisae encontramos retrotransposones de repetición terminal larga (LTR) que se clasifican en un rango de familias que abarcan desde Ty1 hasta Ty5. Después de la transposición, pueden quedar a modo de “cicatrices” LTR residuales (elementos delta) que marcan la zona del genoma donde ha habido un transposón. Aprovechando estas secuencias, se han desarrollado técnicas de amplificación por PCR de regiones “interdelta” para diferenciar entre cepas de S. cerevisiae.
Conclusión
A través de su evolución, las cepas enológicas de S. cerevisiae han adquirido una serie de características genéticas y genómicas que les han permitido predominar en este ambiente, haciéndolas más tolerantes a factores de estrés tanto generales como específicos de la fermentación. El conocimiento de estas adaptaciones es muy relevante para el desarrollo, como cultivos iniciadores, de nuevas cepas enológicas de S. cerevisiae con características alineadas con las demandas actuales del mercado. Esto se puede conseguir mediante la selección de nuevos aislados naturales o mediante técnicas de mejora genética, como la mutagénesis al azar y la hibridación inter o intraespecífica. En cualquier caso, hay que tener en cuenta las interacciones ecológicas de los cultivos iniciadores de S. cerevisiae con otras cepas de levaduras, tanto Saccharomyces como no-Saccharomyces, así como el uso eventual de cultivos iniciadores de otras especies.
Fuente: ACE Enología. Autores: Miguel Mejías-Ortiz y Ana Perea-Martínez del Instituto de Ciencia de la Vid y del Vino-CSIC. Universidad de La Rioja, Logroño, España.